En termes simplistes, les anticorps remplissent deux fonctions principales dans différentes régions de leur structure. Alors qu’une partie de l’anticorps, le fragment de liaison à l’antigène (Fab), reconnaît l’antigène, l’autre partie de l’anticorps, appelée fragment cristallisable (Fc), interagit avec d’autres éléments du système immunitaire, comme les phagocytes ou les composants de la voie du complément, pour favoriser l’élimination de l’antigène.
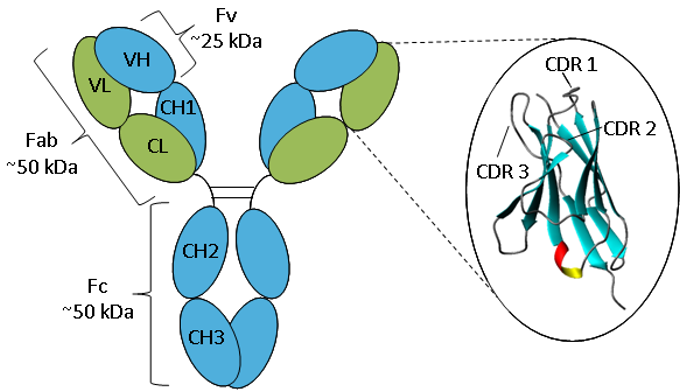
Figure. Représentation schématique d’une IgG.
Un anticorps est constitué de deux chaînes lourdes (bleu) et de deux chaînes légères (vert) repliées en domaines constants et variables. L’agrandissement du domaine variable montre une représentation en ruban de la trame β-sheet et des boucles CDR.
Les anticorps ont tous la même structure de base constituée de deux chaînes lourdes et de deux chaînes légères formant deux bras Fab contenant des domaines identiques à chaque extrémité attachés par une région charnière flexible à la tige de l’anticorps, le domaine Fc, ce qui donne la forme classique en « Y ». Les chaînes se replient en plis répétés d’immunoglobuline constitués de feuillets β antiparallèles (1), qui forment des domaines constants ou variables. Les domaines Fab sont constitués de deux domaines variables et de deux domaines constants, les deux domaines variables constituant le fragment variable (Fv), qui assure la spécificité antigénique de l’anticorps (2), les domaines constants servant de cadre structurel. Chaque domaine variable contient trois boucles hypervariables, connues sous le nom de régions déterminant la complémentarité (CDR), réparties de manière égale entre quatre régions de cadre (FR) moins variables. Ce sont les CDR qui fournissent un site de reconnaissance spécifique de l’antigène à la surface de l’anticorps et l’hypervariabilité de ces régions permet aux anticorps de reconnaître un nombre presque illimité d’antigènes (3).
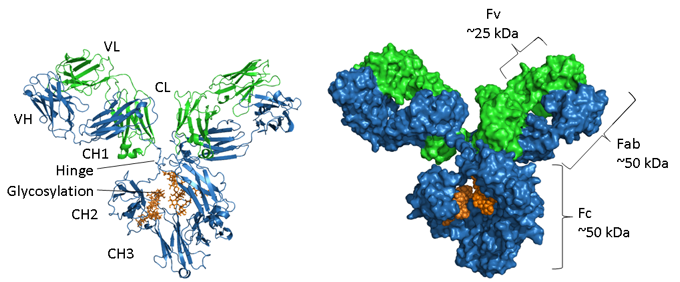
Figure. Représentations structurelles d’une IgG.
La chaîne lourde est représentée en bleu, la chaîne légère en vert et la glycosylation en orange. A gauche, une représentation en ruban montrant les éléments de la structure secondaire et à droite, un modèle rempli d’espace de la même molécule. Le numéro d’accession PDB de l’IgG1 de souris est 1IGY.
Les anticorps sont des protéines glycosylées, la position et l’étendue de la glycosylation variant selon les isotypes. Comme le montre l’image ci-dessus, la région Fc d’une IgG est constituée de deux domaines CH3 appariés et, à l’inverse, de deux domaines CH2 qui sont séparés et n’interagissent pas, mais entre lesquels sont interposées deux chaînes d’oligosaccharides. Ces chaînes couvrent les faces hydrophobes qui conduiraient normalement à l’appariement des domaines. Les N-glycanes contiennent une région centrale commune de deux résidus N-acétyl-glucosamine (GlcNAc) liés à une asparagine (N297 dans les IgG1 humaines) par une liaison amide et trois résidus mannose. Cette structure centrale peut contenir des sucres terminaux supplémentaires, tels que le mannose, le GlcNac, le galactose, le fucose et l’acide sialique, générant une grande hétérogénéité (4).
<< Un bref historique des anticorps Isotypes d’anticorps & sous-types >>
<<. Présentation des anticorps >>
- Poljak, R.J., Amzel, L.M., Avey, H.P., Chen, B.L., Phizackerley, R.P., et Saul, F. (1973). Structure tridimensionnelle du fragment Fab’ d’une immunoglobuline humaine à la résolution 2,8-A. Proc. Natl. Acad. Sci. U.S.A. 70, 3305-3310.
- Inbar, D., Hochman, J., et Givol, D. (1972). Localisation des sites de combinaison d’anticorps dans les parties variables des chaînes lourdes et légères. Proc. Natl. Acad. Sci. U.S.A. 69, 2659-2662.
- Morea, V., Lesk, A.M., et Tramontano, A. (2000). Modélisation des anticorps : implications pour l’ingénierie et la conception. Methods 20, 267-279.
- Wright, A., et Morrison, S.L. (1998). Effet de la structure des glucides associés au C2 sur la fonction effectrice des Ig : études avec des anticorps chimériques souris-humains IgG1 dans des mutants de glycosylation de cellules ovariennes de hamster chinois. J. Immunol. 160, 3393-3402.